- Forschung
- Publikation
Meldung vom:
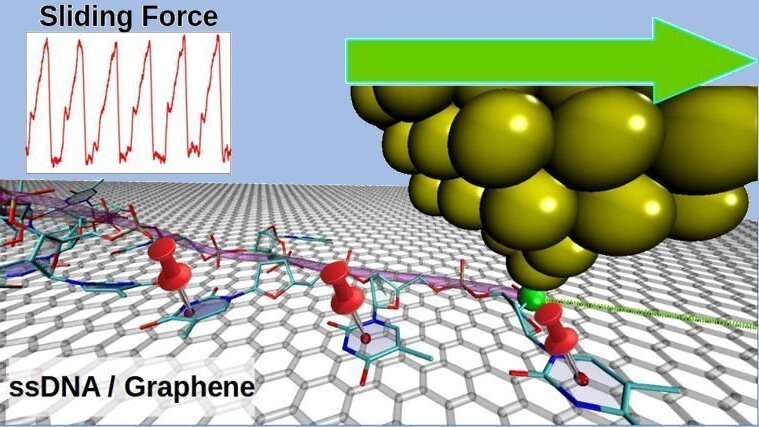
Wir haben molekulardynamische Simulationen von Nanomanipulationsexperimenten an kurzen, einzelsträngigen DNA-Ketten durchgeführt, die auf einer Graphenoberfläche elastisch bewegt wurden. Nach einer kurzen Transiente wurden reproduzierbaren Stick-Slip-Zyklen an Ketten aus 10 Einheiten von Thymin, Cytosin, Adenin und Guanin beobachtet. Die Zyklen haben die Periodizität des Graphensubstrats und finden über eine Zwischenstufe statt, die als Eintauchen in die Sägezahnschwankungen der seitlichen Kraft auftritt, die aufgezeichnet wird, während die Ketten manipuliert werden. Guanin weist bemerkenswerte Unterschiede zu den anderen Basen auf, da in diesem Fall eine geringere Anzahl von Nukleotiden an dem Substrat haftet. Dennoch sind die Größen der statischen Reibung und der lateralen Steifigkeit für alle Ketten ähnlich (30 pN bzw. 0,7 N/m pro adsorbiertem Nukleotid).
Publikation
J. G. Vilhena, Enrico Gnecco, Rémy Pawlak, Fernando Moreno-Herrero, Ernst Meyer, Rubén Pérez: "Stick-Slip Motion of ssDNA over Graphene", Journal of Physical Chemistry B 122 (2018) 840–846, DOI: 10.1021/acs.jpcb.7b06952Externer Link